

Épissage. Un article de Wikipédia, l'encyclopédie libre.
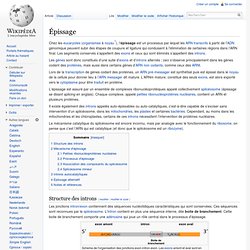
Les gènes sont donc constitués d’une suite d’exons et d’introns alternés ; ceci s’observe principalement dans les gènes codant des protéines, mais aussi dans certains gènes d’ARN non codants, comme ceux des ARNt. L’épissage est assuré par un ensemble de complexes ribonucléoprotéiques appelé collectivement splicéosome (épissage se disant splicing en anglais). Chaque complexe, appelé petites ribonucléoprotéines nucléaires, contient un ARN et plusieurs protéines. Il existe également des introns appelés auto-épissables ou auto-catalytiques, c’est-à-dire capable de s’exciser sans intervention d’un splicéosome, dans les mitochondries, les plastes et certaines bactéries. Cependant, au moins dans les mitochondries et les chloroplastes, certains de ces introns nécessitent l’intervention de protéines nucléaires.
Structure des introns[modifier | modifier le code] Schéma de l'organisation des jonctions exon-intron-exon. Notion de gène morcelé et d'épissage. A – Les données Le gène qui code pour la chaîne bêta de l’hémoglobine est un bon support pour introduire la notion de gène morcelé.
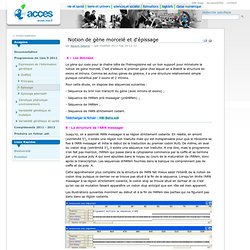
C’est d’ailleurs le premier gène chez lequel on a établit la structure en exons et introns. Comme les autres gènes de globine, il a une structure relativement simple puisque constitué par 3 exons et 2 introns. Pour cette étude, on dispose des séquences suivantes : - Séquence du brin non transcrit du gène (avec introns et exons) ; - Séquence de l'ARNm pré-messager (préARNm) ; - Séquence de l'ARNm ; - Séquence de l'ARN strictement codant.
Télécharger le fichier : HB-Beta.edi B - La structure de l’ARN messager Jusqu'ici, on a assimilé l’ARN messager à sa région strictement codante. Cette appréhension plus complète de la structure de l’ARN fait mieux saisir l’intérêt de la notion de codon stop puisque ce dernier ne se trouve pas situé à la fin de la séquence. Il est aussi possible de le faire avec la fonction dotplot (graphique de ressemblance). e - Schématisations. Étude de la régulation de l’épissage alternatif. Net_efekt (CC BY) Je vais vous exposer dans cet articles un exemple de collaboration entre biologistes et bioinformaticiens afin de répondre à un problème biologique complexe : l'étude de la régulation de l'épissage alternatif.

Je vais vous présenter dans un premier temps le contexte biologique, à savoir ce qu'est l'épissage et comment il est régulé. Puis je vais vous décrire les méthodes de biologie moléculaires permettant de détecter d'une part les événements épissages alternatifs (puces à exons, RNAseq), et d'autre part les liaisons protéines/ARN (iCLIP). Enfin, je vous présenterai les méthodes d'intégration de ces données pour répondre à la question : comment une protéine influence le choix des exons à épisser. (Attention, article très long!) 1.1- L'épissage Figure 1 : Synthèse et maturation de l'ARN messager chez les eucaryotes L'épissage alternatif est un processus biologique découvert à la fi n des années 1970 et constitue l'un des mécanismes les plus complexes chez les eucaryotes.